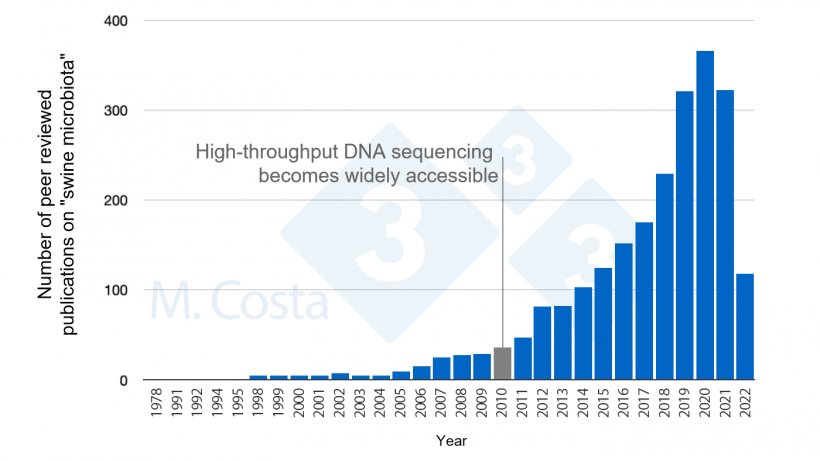
在我们之前的讨论中,我们强调了对猪微生物群进行科学合理的研究是多么具有挑战性。尽管如此,我们已经看到越来越多的关于这个主题的研究(图1)。随着更多信息的出现,你如何才能理解这些数据,并将其应用到猪舍中?
第1步–了解你的条件。
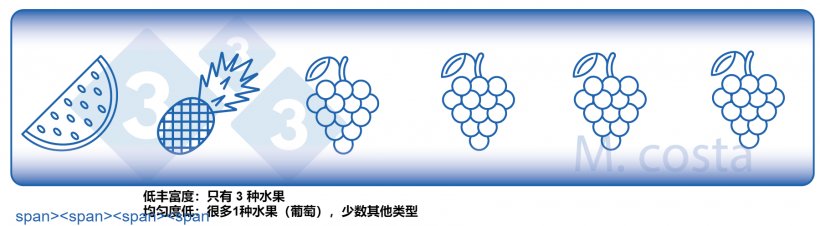
微生物组研究中使用了一些关键指标,有助于我们对结果的理解。 许多都是从“宏观”生态学中借用来的——毕竟,肠道中的细菌群落与大海中的鱼类群落没有太大区别。它们都与环境和栖息地的其他生物有关系。因此,了解这些指标将有助于正确解释有关微生物群的实验。一般来说,这些研究侧重于澄清给定样本中存在/不存在哪些微生物——它们回答了“谁在那里”和“有多少”这一一般问题?给定栖息地(如肠道)中不同种类/类型的微生物通常被称为群落多样性。单一栖息地中的这种物种多样性(如水果或细菌)(如断奶后第3天495号育龄猪的粪便)称为α多样性(图2),并考虑到两个主要方面:

- 物种丰富度:有多少不同类型的细菌/物种。
- 物种均匀度:这些物种的分布(丰度),或者是一种微生物占优势(更丰富)。
有许多指标用于衡量α多样性,最常见的是香农指数、Chao1指数或辛普森指数。每一个都反映了α多样性的不同方面。你会在微生物组研究中经常看到这些指数,反映了样本内的多样性。



在比较两个栖息地的细菌群落(例如,使用抗生素处理与未使用抗生素处理的猪粪便样本)时,我们调查了它们的β多样性——样本之间的相似性。这些群落有很多共同点吗(例如,我们能在粪便样本A和B中找到相同的细菌吗,图3)?它们是相似的还是不同的?同样,有许多指标用于衡量这一点,如Unifrac、Jaccard、Bray curtis差异性等。这些指数考虑了群落多样性的不同方面,例如微生物之间的(系统发育)遗传关系,或每个样本中微生物的丰度。
一般来说,在猪的一生中,肠道微生物的α多样性趋于增加,β多样性趋于减少(图4)。

步骤2–研究是否包括适当的对照?
在微生物组学研究方面,应尽一切努力尽量减少污染物的影响。微生物DNA无处不在(即使微生物已经死亡,它们的DNA也还存在),而当前的DNA测序技术只能检测到非常少量的微生物DNA(如第1部分所述)。因此,研究不仅必须包括适当的实验组(例如,在同一批次中使用抗生素或未使用抗生素的猪,饲喂相同的日粮等),而且还必须包括合适的对照组(空收集管、无样品的测序试剂盒),以帮助识别污染物并将其从分析中去除。如果研究中没有披露这些信息,任何结论都可能存在缺陷。
第三步——生物学意义和P值之外(或者:它真的对动物产生了影响吗?)
微生物群落结构很容易受到干扰。例如,肠道微生物群容易受到日粮、物理空间和抗生素的影响。干预很容易导致群落组成发生重大变化(α和β多样性)。在过去几十年中,为证实这一点,进行了非常重要的工作。然而,生物学意义的证据是关键。微生物做的许多“工作”都是多余的。例如,肠道微生物群的许多成员可以产生相同的短链脂肪酸。因此,微生物群落组成(或“谁在那里”)的变化可能不如他们所做的重要。重要的是要寻找间接证据证明后者受到干预的影响,这可能以不同的方式出现:生长速度、疾病抵抗力、肠屏障功能。这确实取决于研究,但应该报告。
与任何其他尖端技术一样,在将研究从实验室工作转移到猪场时,可能会丢失一些重要方面。随着微生物组调节及其在养猪生产中的应用越来越清晰,我们应该看到新的策略来帮助提高性能和疾病抵御能力。
333团队



