猪繁殖与呼吸综合征(PRRS)至今仍是对养猪业经济影响最大的疾病,其防治还远不能令人满意。更好、更全面地了解PRRSV的变异情况以及监测某一地区/国家/大陆内新菌株的传播情况,无疑将有助于兽医和生产者实施控制和可能的根除计划。从1990年底开始,全世界都可进行PRRSV测序,主要是在北美、欧洲和东南亚。
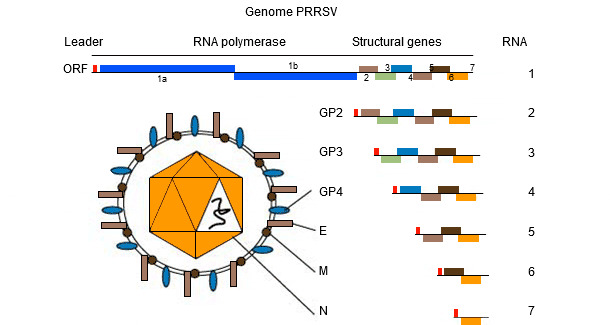
PRRSV基因组(见图1)是单链RNA分子,在宿主复制过程中易发生“错误”(基因突变)。这种“出错倾向”的结果是存在不同的PRRSV菌株,每个菌株都有自己独特的基因序列。这种序列差异是否导致“不同行为”(临床病理学?免疫学?)仍然是从业者和研究人员之间争论的焦点。

测序的基础知识
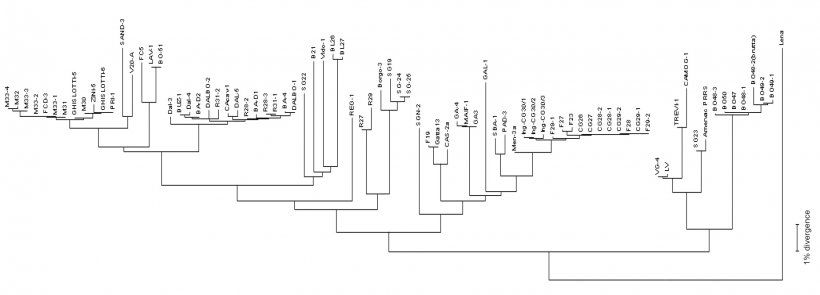
对来自现场样品(血清,组织,口腔液)的PCR产物进行病毒测序,通常是从目标区域(开放阅读框)的一些病毒RNA基因组片段(见图2)获得核苷酸读数,然后使用专用软件进行系统发育分析,比较同源百分比。该过程的结果返回不同PRRSV菌株之间的相似程度(同源性)。使用图形可视化软件,也可以获得一个树形图(或“系统发生树”),显示与参考病毒序列的相关性(或缺乏相关性)(见图3)。

PRRSV基因组编码至少10个ORF。最常用的测序方法是ORF5(编码非糖化蛋白E)和ORF7(编码核衣壳蛋白N),尽管它们分别只占整个基因组的4%和3%。ORF5代表更可变的区域,而ORF7代表更保守的区域。因此,与ORF5相比,在ORF7中发现的相同程度的变异(即5%变异)在遗传变化方面更“戏剧性”。对相似性(即病毒是否相关)的解释需要更多的额外信息,因为基因变化的速度可能是高度可变的。
认真保存所有测序文件是非常重要,以匹配日期、猪场类型(位点1-2-3)、猪生产流程、位置(GPS纬度/经度)和序列的来源(动物/组织/样本的类型)。到目前为止,我们的PRRSV序列数据集包含自2002年起的1300多个ORF7序列。为了解释和理解差异,更重要的是将个体序列与临床事件相匹配,例如流产母猪的数量和在位点1的断奶前死亡率,及在位点2和3的死亡率。
实际问题
生产者和兽医经常问的问题是:
-
序列间观察到的遗传差异是否代表猪场/系统中单个PRRSV菌株的正常变异,或代表猪场/系统中同时存在或在短时间内存在的多个不同菌株?
- 我现在所面临的问题是由“新菌株”引起的“新爆发”,还是再循环?
T为了回答这些问题,我们必须就在一定时间内(12-24个月?)收集到的两种病毒株的可接受的同源性达成一致。换句话说,相似性阈值。97-98%的序列同源性或2-3%的差异通常是可接受的值。根据我的经验,在“临床稳定的封闭群”(传统的母猪群或猪生产流)中,很难看到高于2%的变化,因我们在长达3年的时间里,在一个临床稳定的群体中观察到“同一菌株”。与此相反,每当我们注意到一致的PRRS活性时,就会出现“新的”和系统发育上不同的(90%同源性或更少的)菌株。不幸的是,我们并不完全知道这些我们观察到的巨大差异是否是突然的病毒变化/突变(我个人认为不太可能)或引入新菌株的结果。明确且被广泛接受的事实是,基因相似性/多样性无法预测免疫相似性(即表明存在交叉保护性免疫),且根本无法预测内在致病性(不能说明特定菌株是“好”还是“坏”)。
目前可用的全基因组序列(更多地用于研究目的,而非日常诊断用途)肯定有助于回答这个问题。

根据代表猪场和地区的参考菌株集以及现有商品疫苗的序列分析新的PRRSV序列是非常重要的(这将使区分田间分离株和疫苗株成为可能)。目前,我们仍在使用帕多瓦大学的一套软件来构建我们的系统发育树,并使其在总生产系统中由猪生产流组织起来;我们也可以很快加入另外两个“临时计算机程序”(戴维斯大学加利福尼亚的Bioportal和意大利布雷西亚的IZSLER和CLASSIFARM-PATH),这些程序有更大的序列集来比较,以便更好地理解PRRSV 在意大利和欧盟传播。
感谢Michele Drigo教授对本文进行的修改。
来源:pig333



